-
购物车共件商品总计:
-
询价列表0共0件商品总计:¥0.00
尽管染色质免疫沉淀(ChIP)的数据读取技术在不断地发展,但是其DNA富集的方法中存在的缺点却没有很好的改善。CUT&RUN(Cleavage Under Targets and Release Using Nuclease)作为表观遗传学的研究的一种新型技术方法,对传统的方法进行了重大的修改,以消除ChIP固有的缺点,我们将在下文中详细介绍。
CUT&RUN可用于绘制全基因组转录因子结合位点、染色质相关复合物、组蛋白变体和翻译后修饰的图谱。自该技术面世以来,已有多篇CNS文章利用CUT&RUN技术研究蛋白-DNA的互作。作为ChIP的高效替代方案, CUT&RUN在蛋白质-DNA研究中将会被越来越广泛地使用。
1. 基本原理
CUT&RUN在细胞内利用抗体靶向定位目标蛋白, 再由pA/G-微球菌核酸酶(Micrococcal nucleasel, MNase)对目标蛋白两端的DNA进行切割,从而将目标蛋白质-DNA复合物释放到细胞外。
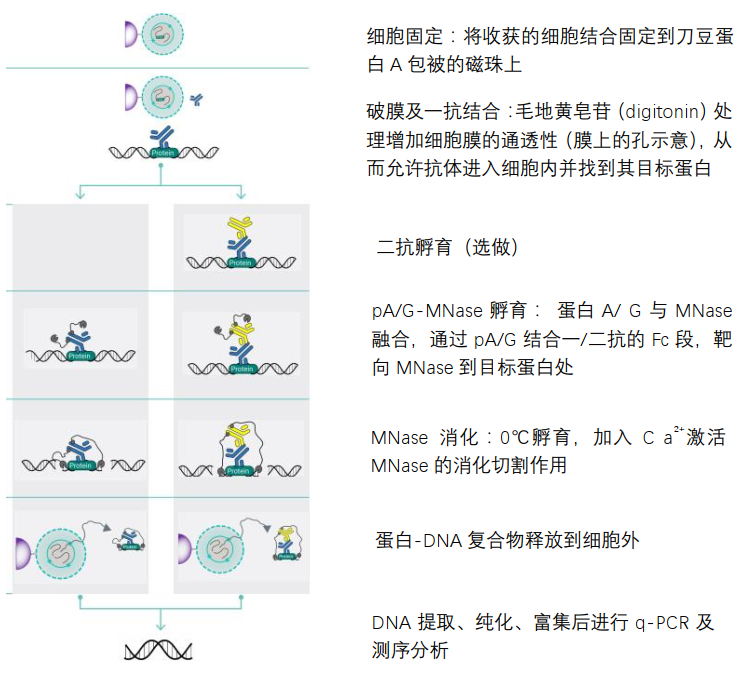
CUT&RUN原理图
2. 产品优势
四正柏提供的试剂盒对原有的CUT&RUN方法进行改进,适用于1-50万个细胞的研究。
本系列产品有以下优势:
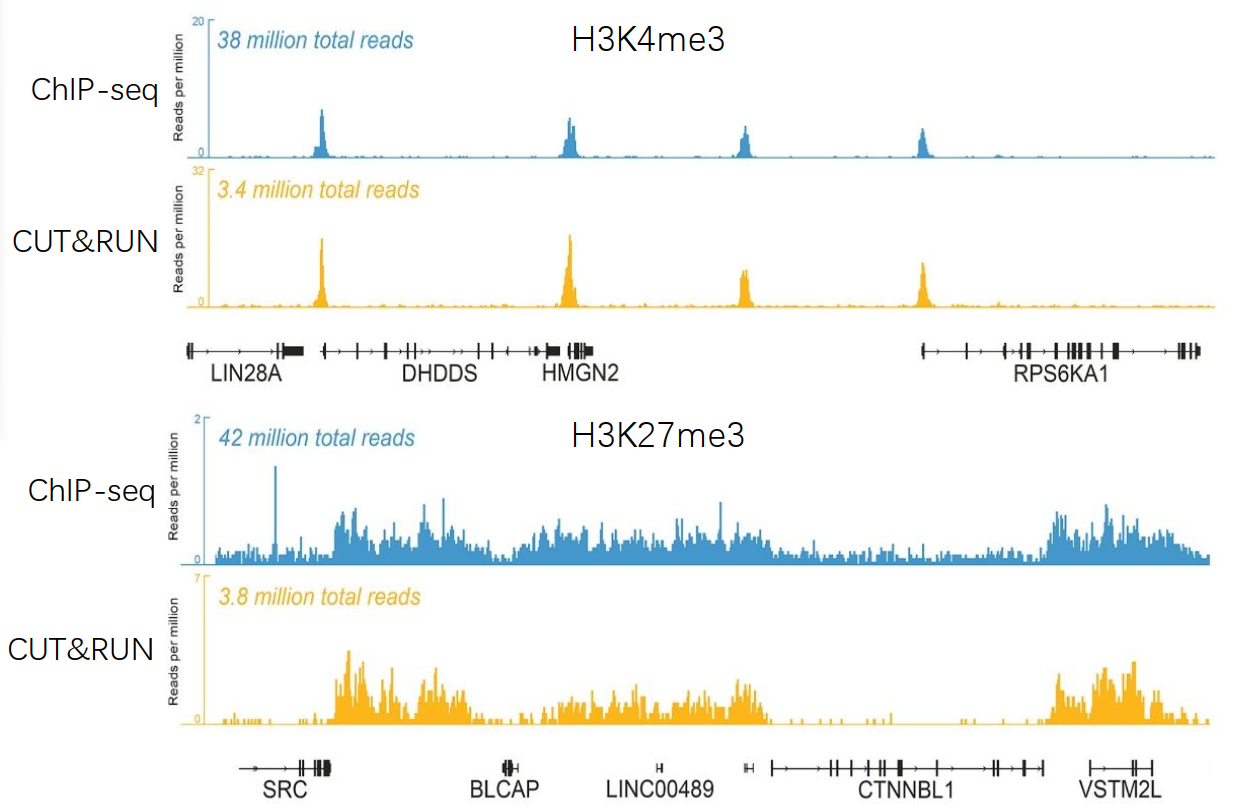
分别对K-562细胞的H3K4me3和H3K27me3所结合的DNA进行测序,用于ChIP的细胞数量为2000万,用于 CUT&RUN的细胞数为50万。对测序结果进行分析,CUT&RUN有着卓越的灵敏度和信噪比,测序深度减少了10倍以上,细胞的需求减少了40倍。
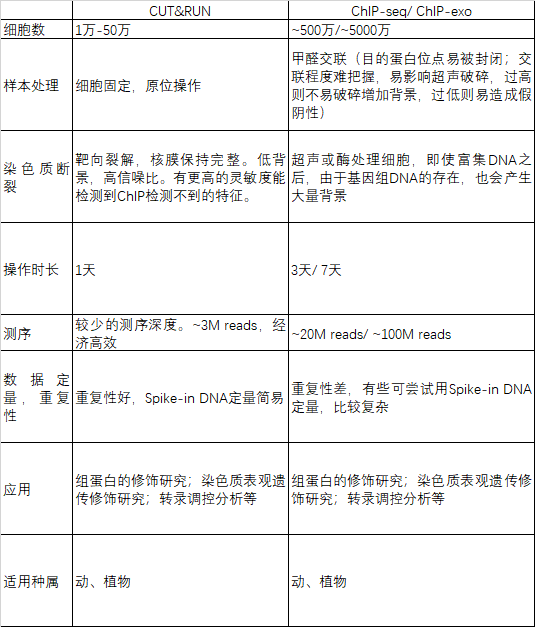
CUT&RUN与Chip-seq对比图
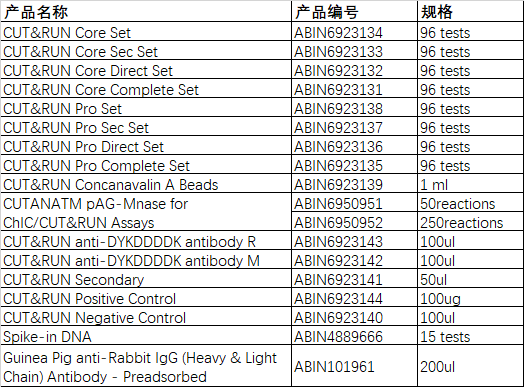
3. 产品信息
常见问题解析
Q:如何验证一抗在CUT&RUN中起作用?
A:对于CUT&RUN实验,验证数据可以包括例如 Tapestation或Bionalyzer图显示大小分布,qPCR数据显示靶标富集。由于CUT&RUN的样本量较低,获得的DNA也相对较少,对于低丰度的靶蛋白,浓度通常太低而无法使用荧光测定法或毛细管电泳测量,所以需要选择高灵敏度的Qubit 或 Nanodrop fluorometer以及Tapestation或Bionalyzer。如果获得的DNA<50bp ,PCR扩增是无法进行的。一旦产生了测序文库并进行了测序图测序,就可以读取并验证读取在已知结合位点的积累。
Q:实验中是否需要使用二抗?
A:二抗的使用取决于抗体和pA/G-MNase融合蛋白的宿主和亚型,对于pA / G-MNase结合,可能是需要的。蛋白A对所有兔IgG抗体具有良好的高亲和力,但对大鼠,山羊和绵羊IgG同种型抗体以及某些小鼠IgG抗体亚类(尤其是IgG1)具有低亲和力。另一方面,蛋白G与小鼠,山羊,绵羊和大多数大鼠IgG的Fc区结合良好。但是,它对兔IgG的亲和力低于蛋白A。当使用我们改进后的CUT&RUN 方案时通常不需要二抗。原始的方案则需要二抗来确保融合蛋白与抗体的有效结合。
Q:CUN&RUN是否可改造适用于RIP-seq?
A:改善CUT&RUN的操作步骤作用在RNA上,以作为RIP-seq的替代方案是有可能可以实现的。细胞质中的RNA如果缺乏5‘cap和3‘poly-A则易被降解,因此建议选用细胞核作为样本。由于核被膜不含胆固醇,所以不需要使用dignitonin。分离出来的细胞核可通过核被膜上的糖蛋白固定在ConA磁珠上,再加入抗体识别目的蛋白,然后加入pA/G-MNase靶向到抗体上,从而切割RNA。最后将RNA分离出来转录成cDNA并进行测序和定位。
更多详情请咨询四正柏生物科技有限公司。
产品经理:刘经理 2853711237
相关文献
引用产品:CUTANATM pAG-MNase for ChIC/CUT&RUN Assays
1. Meers, Bryson, Henikoff, Henikoff.Improved CUT&RUNchromatin profiling tools. eLife, Vol. 8, 2019.
2. Skene, Henikoff, Henikoff. Targeted in situ genome-wideprofiling with high efficiency for low cell numbers. Nature protocols, Vol. 13,Issue 5, pp. 1006-1019, 2019.
3. Skene, Henikoff. An efficient targeted nuclease strategyfor high-resolution mapping of DNA binding sites. eLife, Vol. 6, 2018.
4. Schmid, Durussel, Laemmli. ChIC and ChEC; genomic mappingof chromatin proteins. Molecular cell, Vol. 16, Issue 1, pp. 147-57, 2004.
引用产品 Guinea Pig anti-Rabbit IgG (Heavy & Light Chain) Antibody -Preadsorbed
1. Skene, Henikoff, Henikoff. Targeted in situ genome-wideprofiling with high efficiency for low cell numbers.Nature protocols, Vol. 13,Issue 5, pp. 1006-1019, 2019.
2. Brahma, Henikoff. RSC-Associated Subnucleosomes DefineMNase-Sensitive Promoters in Yeast. Molecular cell, Vol. 73, Issue 2, pp.238-249.e3, 2019.
3. Meers, Bryson, Henikoff, Henikoff. Improved CUT&RUNchromatin profiling tools. eLife, Vol. 8, 2019.
4. Kaya-Okur, Wu, Codomo, Pledger, Bryson, Henikoff, Ahmad,Henikoff. CUT&Tag for efficient epigenomic profiling of small samples andsingle cells. Nature communications, Vol. 10, Issue 1, pp. 1930, 2019.
5. Meers, Janssens, Henikoff. Pioneer Factor-NucleosomeBinding Events during Differentiation Are Motif Encoded. Molecular cell, 2019.
6. Kasinathan, Henikoff. Non-B-Form DNA Is Enriched atCentromeres. Molecular biology and evolution, Vol. 35, Issue 4, pp. 949-962,2019.